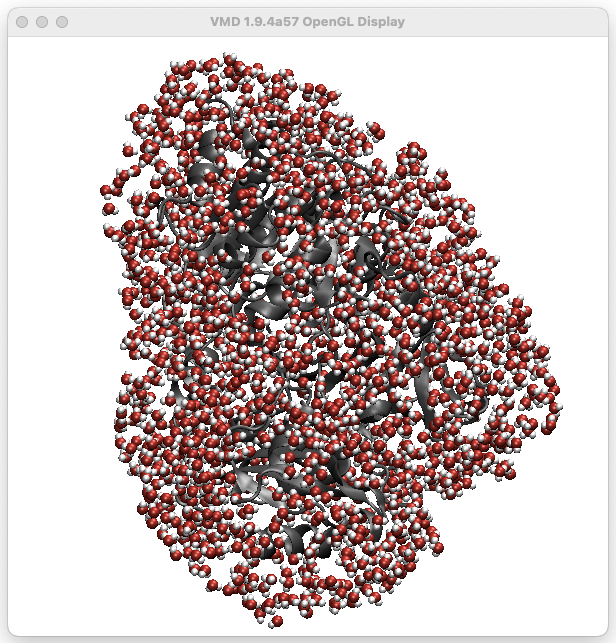
cpptrajでタンパク質周囲5Å以内にある水分子を抽出する
- AmberTools パッケージ に含まれる cpptraj で、特定の分子・残基から一定距離以上のものを削除する方法
手順
- 次のようなシェルスクリプトを書いて、適当な名前(例:
extract.sh)で保存して、実行(sh extract.sh)する
extract.sh
#!/bin/bash
module load amber/22u4
cmd="
parm ../topology.prmtop
trajin trajectory.mdcrd
reference reference.pdb
strip :Na+,Cl-
strip !(:1-541<:5.0)
trajout protein_water.pdb
"
echo "${cmd}" | cpptrajスクリプトの解説
module load amber/22u4- 分子研スパコン
の場合、
moduleでAmberToolsを読み込む
- 分子研スパコン
の場合、
cmd="..."の部分で、cpptrajに渡すコマンドを定義parm ../topology.prmtop- トポロジーファイル
topology.prmtopを読み込む
- トポロジーファイル
trajin trajectory.mdcrd- トラジェクトリファイル
trajectory.mdcrdを読み込む
- トラジェクトリファイル
reference reference.pdb- 距離計算の基準となる参照構造として
reference.pdbを使う
- 距離計算の基準となる参照構造として
strip :Na+,Cl-- ナトリウム(Na+)と塩化物(Cl-)イオンを除去する
strip !(:1-541<:5.0)- タンパク質(例:1〜541番目の残基)から5Å以上の距離にあるすべての原子を除去する
- タンパク質とその周りの5Å以内にある水分子が残る
trajout protein_water.pdb- 結果をPDBファイル
protein_water.pdbに出力する
- 結果をPDBファイル
echoコマンドでcmd変数の内容を出力し、パイプ(|)を使用してその出力をcpptrajに渡して実行する
結果