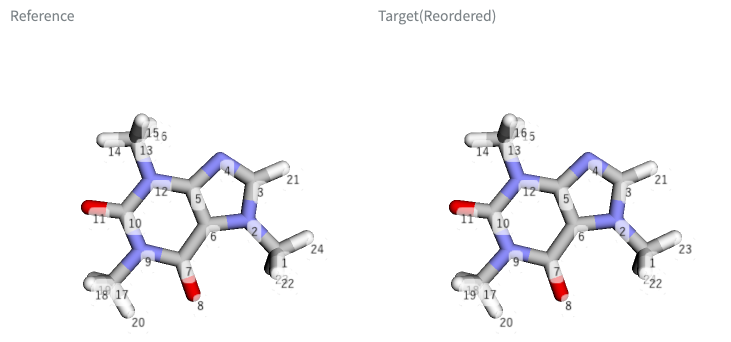
同じ分子なのに原子の並び方が合っていない2つの構造データを適切に並べ替える
はじめに
- 同じ分子についての2種類の異なる構造データがあって、原子の並びが合っていないとき、対応するように、並べ替えをしなくちゃいけない場合がありますよね
- このような並べ替えについて、少し大きなサイズの分子になると手作業では大変なので、自動でやるためのツール(
Atom Aligner
) を作りました
- このツールを使うと、2つの異なる構造を持つ同一の分子データを読み込み、グラフマッチングという方法で原子の順序を適切に並べ替えることができます
- このノートでは、上記のツールを作っているときに調べたことをまとめています
グラフマッチングを用いた原子順序の並べ替え
グラフ
- グラフは、頂点(ノード)とそれらを結ぶ辺(エッジ)で構成されます
- 分子をグラフとして表現する場合、原子を頂点とし、原子間の結合を辺として扱います
分子グラフ
- 分子グラフ $G$ は、頂点集合 $V$ と辺集合 $E$ で構成します
$$G = (V, E)$$
- 各頂点 $v_i$ は原子を表し、頂点の属性として原子の種類 $t_i$ と座標 $(x_i, y_i, z_i)$ を持ちます
- エッジ $e_{ij}$ は、原子間の距離 $d_{ij}$ が閾値 $d_{\text{threshold}}$ 以下の場合に作成します
$$
d_{ij} = \sqrt{(x_i - x_j)^2 + (y_i - y_j)^2 + (z_i - z_j)^2}
$$
$$
e_{ij} \in E \iff d_{ij} < d_{\text{threshold}}
$$
- 今回は、閾値 $d_{\text{threshold}}$ はとりあえず 1.6 Å に設定しています
C-HとC-Cだと長さが違うはずなので、結合の種類ごとに閾値を決めた方がよいけれど、まあ、とりあえずは一定値で
- このようにして作成したグラフは、「原子同士のつながりかた」をもとに、分子の構造を抽象的に表現するものになっています
グラフマッチング
- グラフマッチングとは、2つのグラフ間で対応する頂点と辺を見つけるプロセスです
- 簡単に言うと、2つのグラフが同じ形をしているかどうかを調べ、その形が一致する部分を探します
- 具体的には、2つのグラフ間の同型写像(isomorphism)というものを見つける手続きになります
- 今回の場合、ノードの一致条件として、原子の種類が同じであるものとします
- 2つのグラフ $G_1 = (V_1, E_1)$ と $G_2 = (V_2, E_2)$ が同型であるための条件は、次の写像 $f: V_1 \to V_2$ が存在することです
$$
\forall (v_i, v_j) \in E_1, (f(v_i), f(v_j)) \in E_2
$$
$$
t_{v_i} = t_{f(v_i)}
$$
- 同型写像 $f$ が見つかった場合、そのマッピング情報に基づいて、ターゲットの構造 $G_2$ 内の原子の順序を参照とする構造 $G_1$ に合わせて並べ替えます
- 同型写像の検索は、Python の
NetworkXライブラリにあるGraphMatcherクラスを使うとできます
試してみよう
- 次のように2つの構造データで原子の順番がバラバラになってしまているとき、手作業で並べ替えるのは大変ですよね
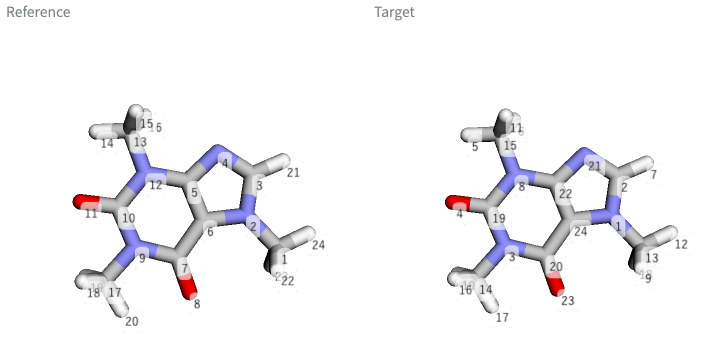
- 今回作った Atom Aligner
を使うと、
Referenceに合うように、Targetとして指定した構造データを自動で並べ替えてくれます- ただし、メチル基にある3つの水素原子など、グラフ表現で区別できないものについて、「見かけが合うように」並べ替えることは(このツールでは)できません